chopping-substrings
— 焉知非鱼第18周挑战赛的第一个任务是在一组字符串中找出最长的共同子串,即给定一组字符串如 “ABABC”、“BABCA “和 “ABCBA”,打印出 “ABC”。
“最佳实践"解决方案是一种被称为后缀树的技术,它需要一些中等复杂的编码。然而,我们可以使用一种简单得多的方法,对长达几十万字符的字符串获得非常合理的性能。
让我们从一个非常简洁,但次优的解决方案开始。如果我们有一个每个字符串的所有可能的子串的列表:
ABABC: ABABC, ABAB, BABC, ABA, BAB, ABC, AB, BA, BC, A, B, C BABCA: BABCA, BABC, ABCA, BAB, ABC, BCA, BA, AB, BC, CA, B, A, C ABCBA: ABCBA, ABCB, BCBA, ABC, BCB, CBA, AB, BC, CB, BA, A, B, C
…那么我们可以把每个列表当作一个集合,取这些集合的交集:
ABC, AB, BA, BC, A, B, C
…然后只需找到最长的元素: ABC
当然,有时所有可能的子串的交集会有一个以上的最长元素:
SHAMELESSLY NAMELESS LAMENESS
…所以我们需要确保我们的算法能够找到所有的子串。
鉴于 Raku 语言中内置了集合和集合操作,这里唯一的挑战就是为每个字符串生成所有可能的子串的完整集合。而且,鉴于 Raku 有非常复杂的正则表达式匹配,这也不是什么真正的挑战。
在普通的 regex 匹配中:
my $str = '_ABC_ABCBA_';
say $str.match(/ <[ABC]>+ /);
# Says: 「ABC」
字符串被搜索为符合模式的第一个子串:在上面的例子中,是 ‘A’、‘B’ 或 ‘C’ 中的一个或多个的第一个序列。但是我们也可以通过添加 :g (或 :global) 副词,要求 Raku regex 引擎返回每个匹配的独立子串:
say $str.match(/ <[ABC]>+ /, :g);
# Says: ( 「ABC」 「ABCBA」 )
或者我们可以要求每一个匹配的重叠子串,通过添加一个 :ov(或 :overlap)副词:
say $str.match(/ <[ABC]>+ /, :ov);
# Says: ( 「ABC」 「BC」 「C」 「ABCBA」 「BCBA」 「CBA」 「BA」 「A」 )
或者我们可以通过添加 :ex (或 :exhaustive) 副词,要求所有可能匹配的子串:
say $str.match(/ <[ABC]>+ /, :ex);
# Says: ( 「ABC」 「AB」 「A」 「BC」 「B」 「C」
# 「ABCBA」 「ABCB」 「ABC」 「AB」 「A」
# 「BCBA」 「BCB」 「BC」 「B」
# 「CBA」 「CB」 「C」 「BA」 「B」 「A」 )
当然,最后一种选择正是我们所需要的:一种提取一个字符串的所有可能子串的方法。除了我们需要找到包含所有可能的字符的子串,而不仅仅是 A/B/C,所以我们需要针对"匹配任何东西"的点元字符进行匹配:
say $str.match(/ .+ /, :ex);
# Says: ( 「_ABC_ABCBA_」 「_ABC_ABCBA」 「_ABC_ABCB」
# 「_ABC_ABC」 「_ABC_AB」 「_ABC_A」)
# ...(54 more elements here)...
# 「BA_」 「BA」 「B」 「A_」 「A」 「_」 )
如上例所示,这些 regex 匹配返回的是一个 Match 对象序列(在输出中用「…」括号表示),而不是一个字符串列表。但这种对象很容易转换为字符串列表,只要对每个对象调用字符串强制方法(使用向量方法调用:"»."):
say $str.match(/ .+ /, :ex)».Str;
# Says: ( _ABC_ABCBA_ _ABC_ABCBA _ABC_ABCB
# _ABC_ABC _ABC_AB _ABC_A
# ...(54 more elements here)...
# BA_ BA B A_ A _ )
为了找到两个或多个字符串的共同子串,我们需要它们每个字符串的详尽子串列表。如果字符串存储在一个数组中,我们可以在该数组上使用另一个向量方法调用("».")对每个字符串进行相同的子串化:
@strings».match(/.+/, :ex)».Str
现在,我们只需要把每一个结果列表当作一个集合,并取它们的交集。这很容易,因为 Raku 有一个内置的 ∩ 操作符,我们可以用它把所有的列表化简成一个单一的交集,就像这样:
[∩] @strings».match(/.+/, :ex)».Str
为了从这个集合中提取一个子串的列表,我们简单地要求它的键:
keys [∩] @strings».match(/.+/, :ex)».Str
我们现在有了一个常见子串的列表,所以我们只需要找到该集合中最长的子串。Raku 有一个内置的 max 函数,但它只能找到第一个最大长度的子串。我们需要所有的子串。
为了得到它们,我们将用一个新的变体来增强现有的内置函数;这个变体将返回每个最大值,而不仅仅是第一个。这个新的子程序与现有的 max 内建函数接受相同的参数,但需要一个额外的 :all 副词,以表示我们需要所有的 maxima。
# "Exhaustive" maximal...
multi max (:&by = {$_}, :$all!, *@list) {
# Find the maximal value...
my $max = max my @values = @list.map: &by;
# Extract and return all values matching the maximal...
@list[ @values.kv.map: {$^index unless $^value cmp $max} ];
}
现在,我们可以只保留最长的普通子串(即那些 .chars 值最大的子串),就像下面这样:
max :all, :by{.chars}, keys [∩] @strings».match(/.+/, :ex)».Str
最初的挑战规范要求程序接受一个字符串列表作为命令行参数,并打印出它们最长的共同子串,它的样子是这样的:
sub MAIN (*@strings) {
.say for
max :all, :by{.chars},
keys [∩] @strings».match(/.+/, :ex)».Str
}
问题解决了!
除了那些讨厌的生物信息学家。给他们这个解决方案,他们会立即在半打 DNA 序列中寻找上万个碱基对中最长的共同子串。并立刻发现这种方法的根本缺陷:长度为 N 的字符串中可能的子串数量为 (N²+N)/2,因此计算和存储它们的成本很快就会变得非常高。
例如,要找到6个10000碱基的DNA字符串中最长的共同子串,我们需要提取和提炼的子串就超过3亿条。这很快就变得不切实际,尤其是当生物信息学家决定他们对几十万个碱基对的DNA链更感兴趣时,这时最初详尽的子串列表现在至少有300亿个元素的总和。我们显然需要一种更可扩展的方法。
幸运的是,设计一个并不难。在一个N个字符的字符串中,任意长度的子串数量是 O(N²),但特定长度M的子串数量只有 N-M+1。例如在 “0123456789” 这样一个10个字符的字符串中,只有6个5字符的子串。“01234”、“12345”、“23456”、“34567”、“45678 “和 “56789”。
因此,如果我们事先知道最长的普通子串有多长(即M的最佳值),我们就可以只生成和比较该长度的子串(/. ** {$M}/),使用 :overlapping 模式匹配(而不是 :ex exhaustive):
@strings».match(/. ** {M}/, :ov)».Str
这样一来,算法的计算复杂度就会降低到一个更可扩展的 O(N)。
当然,最初我们并不知道最长的普通子串会有多长(除了它必须在1到N个字符之间)。所以我们就像所有优秀的计算机科学家在这种情况下所做的那样:我们猜测。但优秀的计算机科学家不喜欢把它叫做 “猜测”,那听起来一点也不科学。所以他们给这种方法起了一个花哨的正式名称:二进制搜索算法(Binary-Chop Search Algorithm)。
在二进制搜索中,我们从某种合理的最佳子串长度的上界和下界($min 和 $max)开始。在每一步中,我们通过平均这两个界限($M = round ($min+$max)/2)来计算我们对最佳长度的"猜测”,然后以某种方式检查我们的猜测是否正确:在这种情况下,通过检查字符串是否真的有长度为 $M 的公共子串。
如果猜测太小,那么最优的子串长度一定更大,所以猜测成为我们新的下界($min = $M + 1)。同理,如果猜测太高,那么它就成为我们新的上界($max = $M - 1)。我们重复这个猜测过程,每一步都将搜索区间减半,直到我们要么猜对,要么用完候选者……当 $min 的更新值最终超过 $max 的更新值时,就会出现这种情况。
因为二元切分搜索会将每一步的搜索间隔减半,所以在最坏的情况下,完整的搜索总是需要 O(log N) 个猜测,所以如果我们用 O(N) 个固定长度的公共子串计算来检查每个猜测,我们最终会用 O(N log N) 个算法来寻找最长的公共子串。
这看起来像这样:
sub lcs (@strings) {
# Initial bounds are 1 to length of shortest string...
my ($min, $max) = (1, @strings».chars.min);
# No substrings found yet...
my @substrs;
# Search until out-of-bounds...
while $min ≤ $max {
# Next guess for length of longest common substring...
my $M = round ($min+$max)/2;
# Did we find any common substrings of that length???
if [∩] @strings».match(/. ** {$M}/,:ov)».Str -> $found {
# If so, remember them, and raise the lower bound...
@substrs = $found.keys;
$min = $M+1;
}
else {
# Otherwise, lower the upper bound...
$max = $M-1;
}
}
# Return longest substrings found...
return @substrs;
}
要达到完全相同的结果,代码量是原来的十倍,但有了它,我们在性能上也有了相当大的提升。单行道解决方案从6个1000字符的字符串中找到最长的共同子串只需要不到90秒的时间;二元切分解决方案在1.27秒内就能找到同样的答案。两种解决方案的总体性能是这样的:
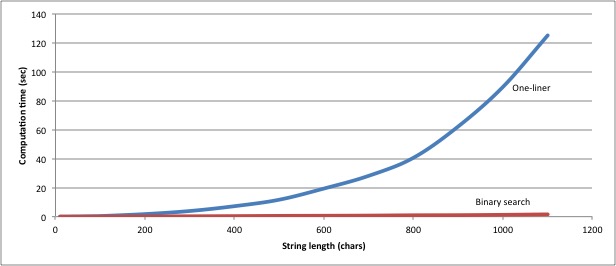
更重要的是,每串1000个字符左右时,单行本就开始失效了 (可能是由于内存问题),而二进制切分版在10000字符以下的字符串都没有问题。
只不过,这时,二元斩方案要花近一分钟的时间来寻找常见的子串。是时候优化我们的优化了。
在较长字符串上的减速似乎主要是来自于使用了一个 regex,在每次迭代时将字符串分解成重叠的M字符子串。Raku 中的 regex 是非常强大的,但它们仍在进行内部优化,所以它们还没有像 Perl 中的 regex 那样快。
更重要的是,在这样的情况下,Raku regexes 有一个基本的性能限制:每个匹配结果都必须以完整的 Match 对象返回,然后再转换回一个常规的 Str 对象。这大大增加了算法运行所需的时间和空间。
但如果我们能直接使用字符串工作,我们就能节省大部分空间,以及大量用于构造和转换 Match 对象的时间。而且,在这种情况下,这很容易做到…因为我们在每个搜索步骤中所需要的就是构造一个固定长度 $M 的重叠子串列表。
“ABCDEFGH” # $str
“ABCDE” # $str.substr(0,$M) “BCDEF” # $str.substr(1,$M) “CDEFG” # $str.substr(2,$M) “DEFGH” # $str.substr(3,$M)
换句话说,对于每个字符串,我们只需要构造:
# For each offset, start at offset, take substring of length M
(0..$str.chars-$M).map(-> $offset { $str.substr($offset, $M) })
…对于所有的字符串来说,它只是:
# For each string...
@strings.map(-> $str {
# Build a list of substrings at each offset...
(0..$str.chars-$M).map(-> $offset {$str.substr($offset,$M)})
})
或者我们可以写得更紧凑,用主题变量 $_ 代替 $str,用占位变量 $^i 代替 $offset:
@strings.map:{(0 .. .chars-$M).map:{.substr($^i,$M)}}
使用该表达式来替换重叠的 regex 匹配,并通过使用内部状态变量而不是外部 my 变量来整理二进制搜索循环,我们得到:
sub lcs (@strings) {
# Search...
loop {
# Bounds initially 1 to length of shortest string...
state ($min, $max) = (1, @strings».chars.min);
# When out-of-bounds, return longest substrings...
return state @substrs if $max < $min;
# Next guess for length of longest common substring...
my $M = round ($min+$max)/2;
# Did we find common substrings of that length???
if [∩] @strings.map:{(0.. .chars-$M).map:{.substr($^i,$M)}}
{
# If so, remember them, and increase the lower bound...
@substrs = $^found.keys;
$min = $M+1;
}
else {
# Otherwise, reduce the upper bound...
$max = $M-1;
}
}
}
改变我们提取候选子串的方式,在性能上又有了很大的提升。基于 regex 的版本需要54.6秒才能找到6个10000字符字符串中最长的共同子串,而这个基于子串的新版本只需3.1秒就能找到相同的答案。而且我们的新版本的扩展性也更好。
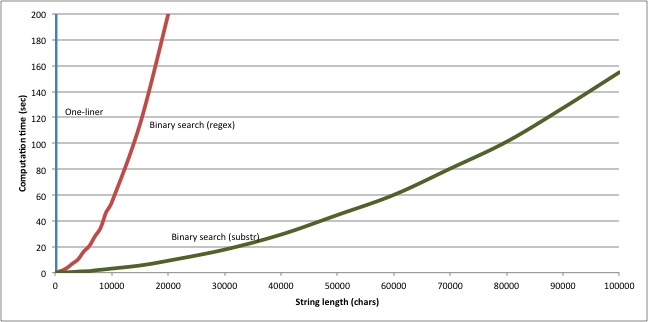
这正好说明:无论你的编程语言有多酷多强大,无论它让你在狡猾的一言一行中实现了多少目标,当涉及到可扩展的性能时,没有什么比正确的算法更好。
by Damian